실제 PCA의 주요 과정
Chapter 6 : Key steps for PCA in Practice
Key steps for PCA in Practice
10.4와 10.5에 걸쳐 PCA를 적용하기 위해 고려해야 할 사항들을 살펴보았다. 이번 장에서는 실제로 PCA를 수행할 때 어떤 단계에 걸쳐 수행하는지에 대해 살펴보자.
-
Mean subtraction
각각의 data 차원에 대해 평균값을 빼준다. 이론상으로는 필요하지 않은 작업이지만, 연산량을 줄이고, 연산 가능 범위를 벗어나는 등 계산하는 데 있어서 발생할 수 있는 문제를 방지해준다
mu = np.mean(x, axis=0) x = x - mu -
Standardization (=scaling)
각각의 data 차원에 대한 standard deviation $\sigma_d$를 나눠준다. 이미지 데이터의 경우엔 각 pixel이 같은 scale을 갖기 때문에 크게 상관없지만, dimension이 각각의 서로 다른 척도 (e.g., 키=m, 체중=kg)로 측정된 경우가 많기 때문에 unit에 대한 편향을 줄이기 위해서 standardization을 수행한다.
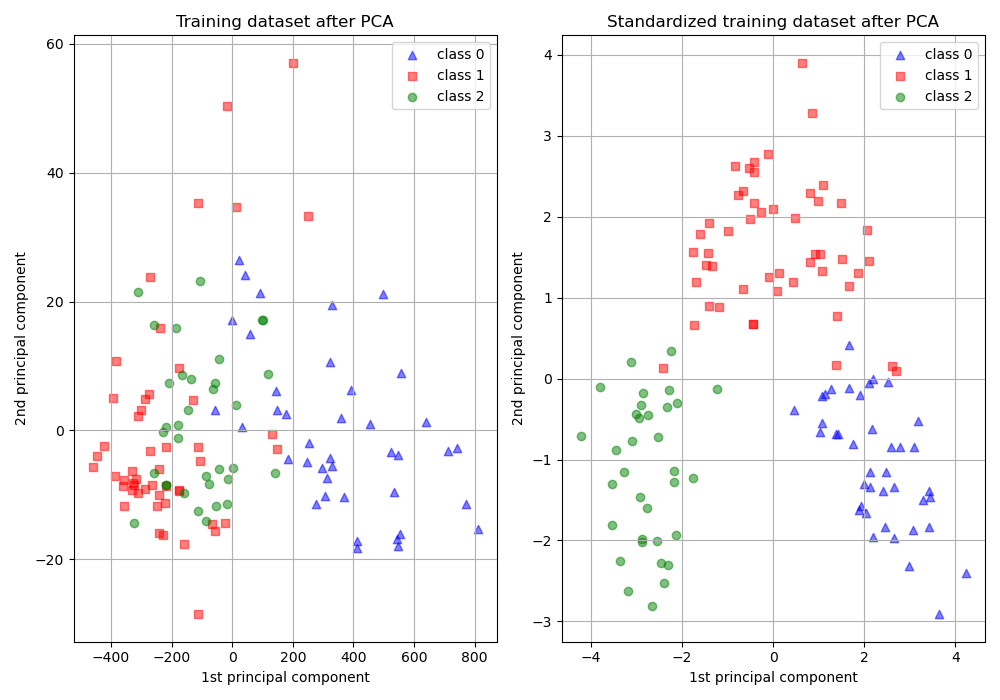
sigma = np.std(x, axis=0) x = x / sigmaStandardization을 수행하지 않은 데이터는 아래 [그림8]과 같이 전혀 다른 결과를 나타낼 수 있다.
### [그림 8] Example of unstardardization(left) vs. standardization(right)
-
Eigendecomposition of the covariance matrix
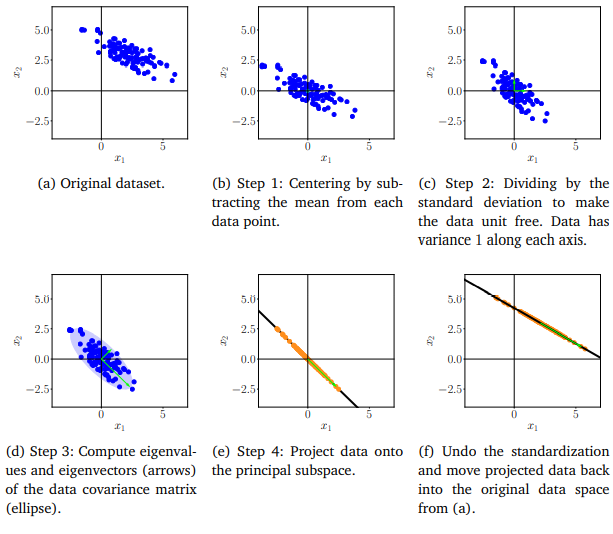
공분산행렬 $S$에 대한 eigenvalues 및 eigenvectors를 구한다. 아래 [그림9] (d)에서는 visualization을 위해 eigenvector에 eigenvalue를 곱한 값을 plot하였으며, $S$를 파란색 타원으로 표현하였다.
# Eigendecomposition S = np.dot(x.T, x)/x.shape[0] eigenvalues, eigenvectors = np.linalg.eig(S) # D개의 eigenvalue가 나옴 # SVD U, D, eigenvectors = np.linalg.svd(x) eigenvalues = np.square(D)/x.shape[0] # N개의 eigenvalue가 나옴 -
Variance Ratio 분석
Eigenvalue를 정렬하고, $\sum_{i=1}^M \lambda_i > T$ ($T$는 임계값)을 만족하는 $M$을 찾는다. (np.linalg.eig 및 np.linalg.svd는 이미 정렬되어서 출력된다.)
eigenvalues_ratio = eigenvalues / np.sum(eigenvalues) cumulative = np.cumsum(eigenvalues_ratio) M = np.argmax(cumulative > T) -
Projection
Standardized data $x_{*}$를 eigenvector위로 투영한다.
B_trunc = eigenvectors[:,:M] x_proj = np.dot(x, B_trunc) -
Decode
Step 1과 2에서 standardization하면서 변형되었던 부분을 다시 원래 상태로 되돌려 놓는다.
x_tilde = x_proj * sigma + mu### [그림 9] Key Steps of PCA in practice